Tento projekt se zabývá oblastí výzkumu neurosvalových onemocnění a různých druhů svalové dystrofie. Neurosvalové onemocnění je obecný pojem pro skupinu poruch (více než 200), které snižují svalovou činnost, a to buď přímo prostřednictvím svalové patologie (svalové dystrofie) nebo nepřímo prostřednictvím nervové patologie. Většina z těchto poruch je vzácná (postihují méně než jednu osobu z 2000), mají genetický původ (80 %) a mezi postiženými jsou jak děti, tak i dospělí. Tato chronická onemocnění vedou ke snížení svalové síly a působí vážná postižení motorických funkcí (pohybu, dýchání, srdce atd.). I přes pokroky v medicíně v současné době není k dispozici žádná léčebná metoda pro osoby postižené neurosvalovými onemocněními.
Tento výzkum do distribuovaných výpočtů přinesli výzkumní pracovníci podporovaní organizací Decrypthon, sdružující AFM (French Muscular Dystrophy Association), CNRS (French National Center for Scientific Research), Universite Pierre et Marie Curie, ENS Lyon, Université Paris XI, Bordeaux 1, Lille 1 a IBM. Zkoumají vzájemné interakce mezi více než 2200 známými strukturami bílkovin se zaměřením na ty, které hrají roli v neurosvalových onemocněních. Vytvořená databáze získaných informací pomůže výzkumníkům navrhnout molekuly, které by mohly vést k léčbě svalové dystrofie a dalších neurosvalových onemocnění.
Ukázka grafického výstupu projektu HCMD při zpracování na počítači.
Jak probíhá zpracování dat v podprojektu HCMD?
Hledání párů vzájemně se ovlivňujících bílkovin a ligandů (ligand je atom, iont nebo molekula, která poskytuje jeden nebo více elektronových párů centrálnímu atomu), nebo větších komplexů vzájemně se ovlivňujících bílkovin patří do základního výzkumu v biologii s bezprostředním dopadem na vývoj nových léků. HCMD je multidisciplinární projekt, který se zabývá právě tímto druhem výzkumu. Zpracování dat spočívá v prohledávání databáze známých bílkovin (obsahující tisíce bílkovin) se zaměřením na určitou oblast vazeb na jiné bílkoviny nebo ligandy. Hlavním úkolem je určit, zda jsou dvě vybrané bílkoviny potenciálními partnery vzájemného působení v buňce. Dále zjišťuje, jak bude tato interakce pozměněna přírodními nebo umělými mutacemi buď v jednom z interagujících partnerů, nebo výsledkem působení vnější molekuly, tedy nějakého farmaceutika. Databáze těchto informací má v medicíně obrovský význam, protože podobný výzkum v laboratorních podmínkách je příliš nákladný a zdlouhavý.
Molekulární modelování za pomoci výpočetní techniky dnes dokáže napodobovat chování molekul na základě různých vstupních dat a ověřovat tak mnoho vědeckých teorií. Vzájemné působení bílkovin a ligandů je technika molekulárního modelování, která vychází z předpovědi polohy a orientace bílkovin ve 3D prostředí. Tato metoda je založena na čistě fyzikálních principech a je schopna do modelů zahrnout i bílkoviny s dosud neznámými funkcemi. Jedinou podmínkou je, že jejich 3D struktura byla určena buď experimentálně nebo ji lze odhadnout na základě některých podobností. Program, který byl vytvořen pro zjednodušení tohoto 3D modelování se nazývá Joint Evolutionary Trees (dále jen JET).
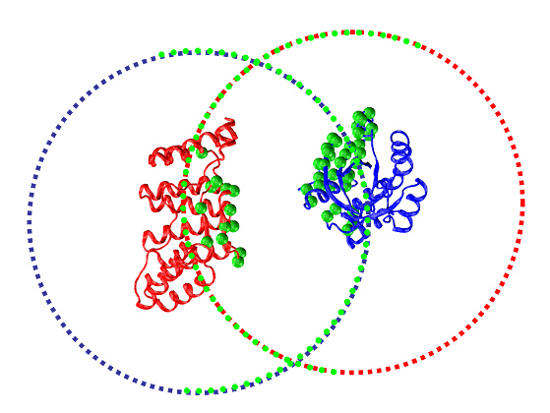
Ukázka funkce programu JET, který systematicky určí výchozí pozici bílkovin, a určí výchozí body pro zpracování (zelené koule na bílkovinách). Modrými a červenými tečkami je znázorněna pozice ligandu. Zelenou tečnou jsou opět zobrazeny ty pozice, které je potřeba zpracovat.
U složitých struktur, jako jsou bílkoviny (nejmenší jsou složeny ze stovek atomů), je k určení správné polohy a vzájemného působení dvou bílkovin zapotřebí obrovského množství výpočetního výkonu. Bez zapojení do distribuovaných výpočtů projektu World Community Grid by výpočetní nároky potřebné k tomuto modelování byly neúnosně náročné. Jen pro zpracování prvních 168 vybraných bílkovin byl odhadovaný čas na 2 Ghz procesoru 8000 let. Lidem trpícím neurosvalovými onemocněními tak díky projektu HCDM svitla naděje, že se svého léku dožijí.
První fáze projektu HCMD
První fáze zpracování dat byla ukončena v červnu 2007. Údaje vyplývající z této první etapy byly jednak použity k nastavení různých parametrů programu JET a také ke zjištění, jak daleko můžeme jít ve filtrování bílkovin pro další zpracování bez ztráty důležitých kandidátů pro pomoc v oblasti neurosvalových nemocí.
Celkem bylo modelováno vzájemné působení 168 bílkovin, které byly zkoumány již pomocí krystalografie a o kterých existovaly předem důkazy o vzájemném působení v buňce. Průzkum probíhal za pomoci algoritmu MAXDo. Byly postupně zkoumány všechny páry bílkovin (28 224) a jejich energetické hodnoty. Dle výsledků samotného provozu první fáze došlo i k menším úpravám algoritmu MAXDo, aby výsledky druhé fáze byly přesnější, zpracování rychlejší a tedy i celkový výzkum efektivnější. Nově upravený algoritmus by měl již automaticky rozlišit výsledky na pozitivní (došlo ke vzájemnému působení bílkovin) a negativní (u kterých ke vzájemnému působení nedošlo). Po zpracování výsledků z první fáze a dokončení příprav byla dne 13. 5. 2009 spuštěna druhá fáze projektu.
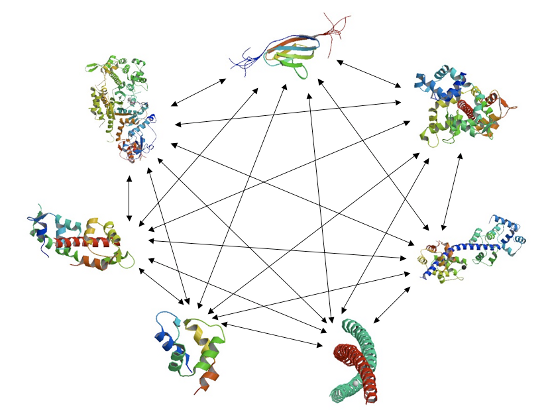
Ukázka vzájemných kombinací všech vybraných bílkovin.
Druhá fáze projektu HCMD
Nová verze MAXDo již zohledňuje informace vytvořené programem JET. Tím došlo k poklesu potřebného výpočetního času k určování výchozí pozice bílkovin o 85 %. V kombinaci s dalšími vylepšeními algoritmu MAXDo je pro modelování vzájemného působení dvou vybraných bílkovin potřeba jen 3% výpočetního výkonu oproti modelováním v první fázi.
Ve druhé fázi výzkumu je zpracováváno celkem 2246 lidských bílkovin, jejichž trojrozměrné struktury jsou známé (a uložené v databázi bílkovin www.rcsb.org). Druhá fáze projektu HCMD tedy zpracovává celkem 2 466 753 párů bílkovin a zkoumá jejich vzájemné působení. Na tento výzkum je tedy i přes velké urychlení výpočtů potřeba několikanásobně více výpočetního výkonu než tomu bylo v první fázi. Čím více výkonu běžných osobních počítačů tedy bude do HCMD zapojeno, tím rychleji půjde výzkum potřebných léků proti neurosvalovým onemocněním a svalové dystrofii kupředu.
Zdroje: World Community Grid
Překlad: forest
Korektura: JardaM